编者的话·写在前面
大自然已经发展出了一系列错综复杂的由蛋白质装配而成的分子机器,这些机器一起完成了维持生命的各项化学反应,从DNA和RNA合成,到蛋白质折叠和翻译后修饰——天然的蛋白质机器展现出了卓越的特异性和协调性。尽管这些蛋白质机器构造复杂,合成生物学家已经成功从多个方面对这些分子机器进行再创。一些经过重新配置的蛋白质机器已经在实验室中诞生,其中的代表是能够合成非传统遗传聚合物 (alternative genetic polymer) 的DNA/RNA聚合酶,以及能够在蛋白质中高效加入非天然氨基酸 (unnatural amino acids, UAAs) 的核糖体。这些成果为人类在未来制造更加复杂的生物大分子机器提供了巨大的想象空间,包括用基因编码指导组装的纳米结构材料,具有生物正交性的化学探针与蛋白质官能团的共翻译组装,用全新的非天然的化学材料组装新的蛋白质等等。
对天然蛋白质机器的设计、改造也促进了合成生物学家们在人工设计细胞 (designer cell) 发展上的投入。科学家们希望在将工程化后的、经过刻画的生物元件 (biological parts) 组装成为功能系统或者人工设计细胞后,能够对其功能和行为进行预测。然而,这之中有一个关键的技术难点,就是要创造一个稳定的、多层的基因调控网络来准确控制系统中各种组分,例如各类蛋白质机器的组装和功能。如今,科学家们已经在开发各种稳健的、可调的RNA类和蛋白质类的调控因子上取得了一系列进展,再加上我们已有研究和刻画的其他正交蛋白质机器——例如RNA聚合酶和核糖体,这些成果也为我们构造和组装人工生物网络系统打下了基础。
对自然界中广泛存在的各种蛋白质分子机器进行工程化改造和再创将会为新生物技术的开发创造广泛且巨大的价值。但是,对蛋白质分子机器的机理、功能和它在整个生物系统网络中的作用进行系统的研究是相当复杂的,需要物理学、化学、工程学,甚至各种尚未诞生的方法学的支持。幸运的是,合成生物学家们跨越了不同学科的界限,并且在改造这些蛋白质机器用于新的生物学功能上已经取得了一些可喜的成果。在本文中,笔者将会介绍在合成生物学和生物技术领域中人们理解、设计和改造蛋白质机器所取得一些关键的、具有代表性的进展。
作者 | 张益豪
编辑团队 | 罗训训 孟凡康 孙智 张益豪
01. 改造DNA合成机器,新型核酸聚合物的合成
DNA聚合酶(DNA polymerase,DNA Pol)是一类在细胞中负责遗传物质的复制和扩增的蛋白质机器,对DNA进行直接操作或者进化的各类技术都离不开DNA聚合酶。所以DNA聚合酶是目前为止被工程化改造最多的一类蛋白质机器。在我们现有的DNA聚合酶工具箱中,包括了用于易错 PCR (error-prone PCR) 的缺乏3’->5’外切酶活性的DNA聚合酶,引入增强持续合成能力结构域从而提高速度和保真度的DNA聚合酶,具有泛底物特异性允许掺入修饰碱基(例如带有荧光标记的dNTP)的DNA聚合酶,等等。
那么我们如何得到具有各种独特功能的DNA聚合酶呢?2001年,Philipp Holliger组的研究者们开发了称为“区分性自我复制” (compartmentalized self-replication,CSR) 的通用的选择策略,极大地促进了新功能DNA聚合酶的开发。研究者利用DNA聚合酶扩增DNA的固有能力来扩增自己的DNA序列,将表型和基因型关联了起来。具体来讲,就是将DNA聚合酶的组合文库在大肠杆菌中进行过表达,然后将细菌文库转移到油-水乳液中。在热处理分解细胞结构后,每个细胞所包含的聚合酶和其模版被保留在乳液的小泡中。在设定的各种选择条件下,符合所需表型的DNA聚合酶变体在一轮轮反应中逐渐脱颖而出。CSR方案已经被用于生产许多具有独特特性的DNA聚合酶,这些新特性包括高热稳定性,能应对复杂核酸结构,拥有更强的DNA结合能力,对抑制子不敏感等。
在CSR方法的基础上,合成生物学家在改造聚合酶取得了更多的进展。Philipp Holliger组的研究者们基于CSR的原理,进一步的开发了一种新的“区分型自我签” (compartmentalized self-tagging, CST) 技术,用于开发各种能够添加异源核酸 (xeno nucleic acids, XNA) 的DNA聚合酶,从而用于制造各种新型的核酸聚合物。如同CSR方法一样,CST法也是在油-水乳液中对聚合酶文库进行筛选。不同的是,这里使用生物修饰的XNA引物来起始PCR反应。此时,能够识别引物发生延伸反应的聚合酶就会结合在质粒模版上,通过链霉亲和素的小磁珠将复制复合物沉淀下来,进入下一轮筛选。
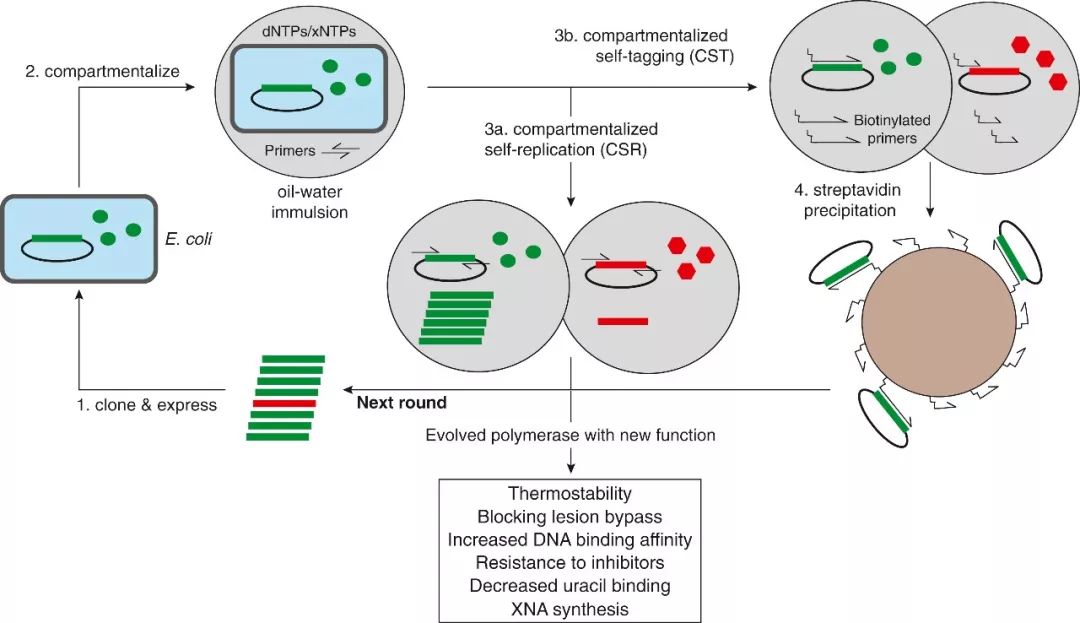 图1.进化得到新特性DNA聚合酶的方法CSR和CST。这些方案已经被用于生产许多具有独特特性的DNA聚合酶,这些新特性包括高热稳定性,能应对复杂核酸结构,拥有更强的DNA结合能力,对抑制子不敏感,以及最终能够合成非天然遗传聚合物。(Glasscock et al.)
图1.进化得到新特性DNA聚合酶的方法CSR和CST。这些方案已经被用于生产许多具有独特特性的DNA聚合酶,这些新特性包括高热稳定性,能应对复杂核酸结构,拥有更强的DNA结合能力,对抑制子不敏感,以及最终能够合成非天然遗传聚合物。(Glasscock et al.)
带有XNA的新型核酸聚合物让科学家们能够对核酸的化学性质进行调整,其中包括螺旋的构象,双螺旋稳定性,甚至改变碱基配对的偏好性(构建G·U摇摆配对)。这些经过改造的特殊化学性质有许多潜在的应用,例如各类基于核酸的新型疗法的开发。
图2.XNA的化学空间图。x轴,y轴和z轴分别代表糖,碱基和骨架的修饰这三个“变量”。大多数已有研究的XNA(PNA除外)都位于轴上,仅对三个“变量”之一进行了修改。通过对XNA空间的更全面的探索,也就是XNA的复制和演变,包括对碱、糖和骨架的修饰组合,我们可以获得更多不同的表型的XNA。(Pinheiro and Holliger)
02. 改造RNA合成机器和多组分遗传系统的控制
RNA合成是基因表达的第一步,也是我们控制细胞行为和基因线路功能的最重要的调节步骤。当我们需要在一个细胞内协调多种组分的活性,开发一套能够对基因表达进行正交控制的工具尤其必要,我们才能对各个基因实现单独的控制而又避免出现互相串扰 (cross-talk)。为了开发这样一套工具,合成生物学家们利用理性设计 (rational design) 结合实验室进化 (laboratory evolution) 的方法对RNA合成机器进行了不少改造工作。
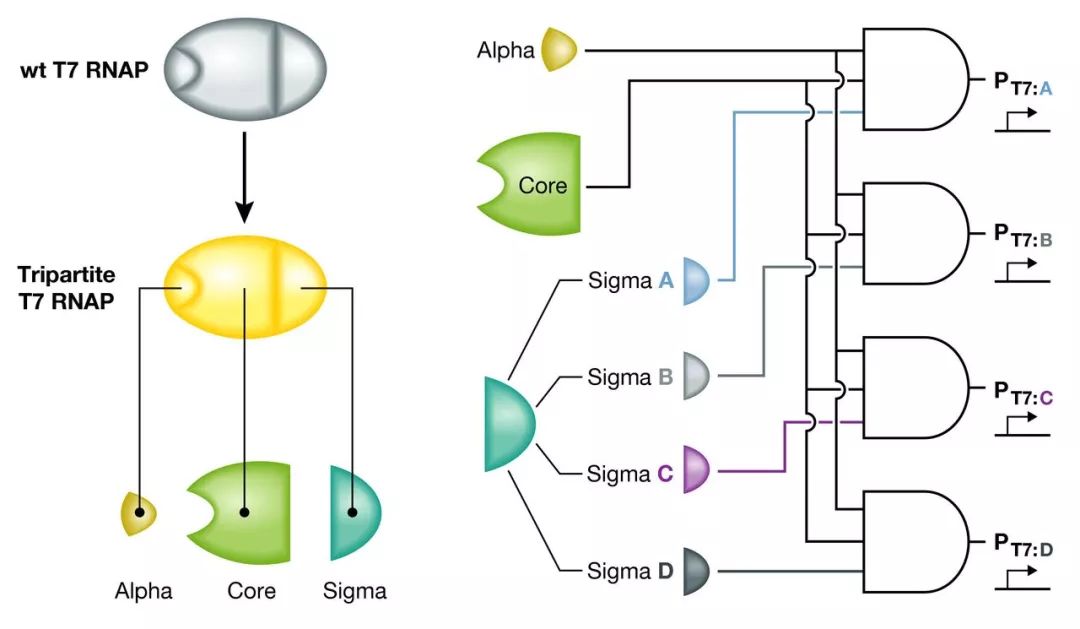
噬菌体T7的RNA聚合酶 (T7 RNA polymerase, T7 RNAP),因为其转录严格可调,并且与宿主基因表达的串扰最小,因此一直以来被用于蛋白表达系统。然而,标准的T7 RNAP表达体系对宿主细胞有较强的毒性,能在单细胞内同时使用的正交的变体也较少。对此,Christopher Voigt组的研究人员通过理性设计的方式开发出了毒性更小的T7 RNAP,并以此为基础衍生出四个具有正交性的T7 RNAP变体。研究人员首先利用定向突变调整T7 RNAP的转录强度和活性,从而降低其在E. coli中的毒性。接着,研究人员利用生物信息学进行元件信息的挖掘,从已有的基因组数据库中找到了多种噬菌体RNAP序列,并进行合成和测试。其中,研究人员发现这些被鉴定出来的噬菌体RNAP在其β发卡结构中存在较丰富的差异,于是他们将这些不同的β发卡插入到已做过突变减毒的T7 RNAP骨架中,构建出了43种新合成的T7RNAP,经过测试筛选最终得到了四种正交的T7RNAP工具。研究人员利用这套工具构建了一个能够在两条代谢通路之间进行切换的AND Gate,展现了这套低毒的转录调控工具用于代谢控制的潜力。
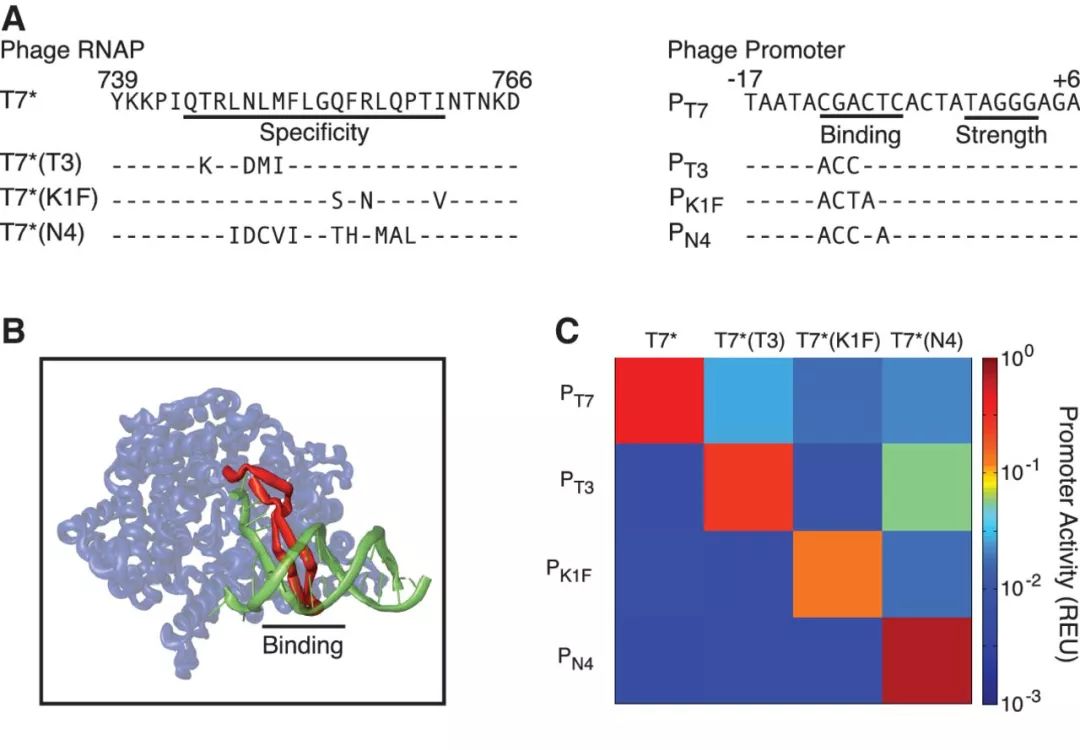 图3.正交T7 RNAP和启动子的设计。A. 改造得到的正交T7 RNAP“结合特异性环”上的氨基酸突变及相应的启动子序列。B. T7 RNAP与启动子序列的结合。C. 改造T7 RNAP的正交性测试。(Temme et al.)
图3.正交T7 RNAP和启动子的设计。A. 改造得到的正交T7 RNAP“结合特异性环”上的氨基酸突变及相应的启动子序列。B. T7 RNAP与启动子序列的结合。C. 改造T7 RNAP的正交性测试。(Temme et al.)
David Shis和Matthew Bennett试图通过另一种思路来简化T7转录系统,那就是对T7 RNAP进行分割,当只有两个片段在细胞内同时表达时,才能重新形成有活性的T7 RNAP。有趣的是,T7 RNAP分割片段不会对细胞产生毒性,并且研究人员对分割片段中决定DNA序列结合特异性的片段进行突变后,得到了三种具有高度正交性的分割T7 RNAP系统。如此一来,我们可以将T7 RNAP的N端片段和C端片段放在不同的启动子后,构建具有不同行为的两输入转录逻辑门。
图4.利用分割T7RNAP构建转录调控的逻辑门。(Shisand Bennett)
在此之后,Christopher Voigt组的研究人员对分割T7 RNAP进行了更进一步的探索,尝试开发一套能够调控分配细胞中有限的T7 RNAP转录机器资源的“资源分配器”。与大肠杆菌RNA聚合酶机器中的σ因子决定了转录过程的调控类似,研究人员通过突变和筛选的方法对T7 RNAP的各个功能片段进行了定位,在67位残基至601位残基可将T7 RNAP分为核心片段,和DNA结合的σ片段,以及一个α片段。调控核心片段可用于控制细胞内T7 RNAP的总体水平,而在不同启动子下控制表达不同的正交σ片段会相互竞争核心片段。通过调整不同σ片段的表达强度,不同受调控基因即可分配到特定比例的T7 RNAP资源。此外,在缺乏α片段时,核心片段与σ片段结合后不会产生转录活性。因此,研究人员可以通过调控细胞内α片段的量来控制整个系统整体的转录强度。同时,在引入了能与正常α片段进行竞争的失活α片段,我们甚至可以独立于质粒拷贝数这样的影响因素,将系统整体的转录强度维持在一个恒定的水平。通常,合成生物学家在将调试好的基因线路在不同的遗传载体上转移后(例如从高拷贝质粒转移到低拷贝质粒或细菌染色体上),常常还需要再进行大量的调试工作。Voigt组这套系统的亮点正是为这一问题提供了一种可能解决方案。也许在将来,我们可以利用这样的一套系统在易于进行操作的质粒上完成各种基因线路的设计刻画,之后直接转移到能够稳定表达的细菌染色体上进行实用,而不再需要重新调试我们的基因线路。
图5.将T7 RNAP分割成三种组分后,与α片段、核心片段共表达不同的σ片段可实现多输入的转录与门网路。(Shisand Bennett)
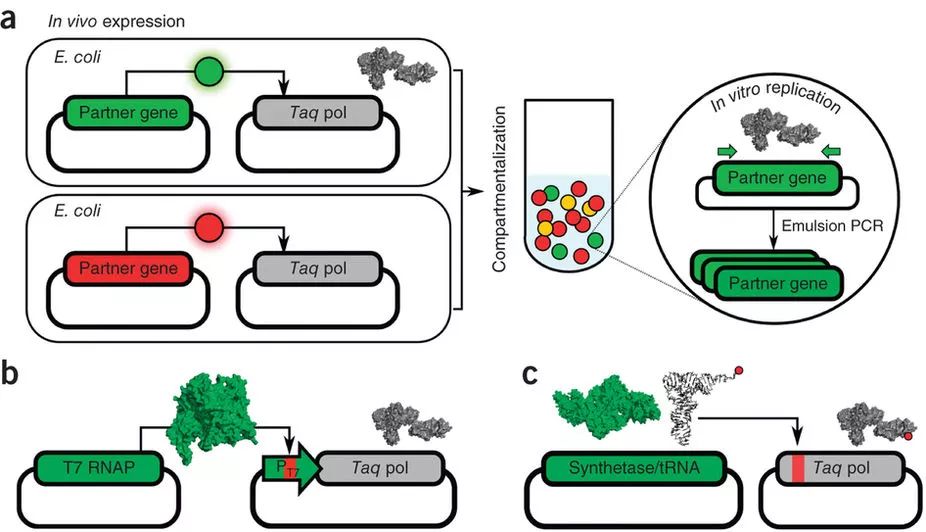
合成生物学家们已经以T7 RNAP为基础开发了多种多样工具,然而,筛选正交的RNA聚合酶需要在细胞内引入较多的外来基因,筛选测试工作复杂。好在,各种新出现的进化方法提供了多种更加高效的方案。其中一种,是Andrew Ellington组开发的另一种基于CSR原理的方法,称为“区分型联合扩增” (compartmentalized partnerreplication, CPR),可用于寻找识别各种正交序列的T7 RNAP。具体的设计是将Taq酶放在包含正交序列的启动子下,识别效果越好的T7 RNAP突变体将使细胞积累越多的Taq酶。在用油-水乳液处理细胞后启动PCR程序,各个小液泡内含有相应T7 RNAP突变体的质粒被Taq扩增。经过几轮重复,对正交位点识别效果最好的T7 RNAP基因将会留存。通过CPR这种方法,研究人员筛选出了一套包含六种T7 RNAP的正交转录系统,这样一套系统可供合成生物学家们设计一个更加庞大复杂的基因线路。
图6.CPR原理示意图。除了筛选正交T7RNAP,该方案还可用于筛选文库中具有特定功能的tRNA/tRNA合成酶(例如可识别UAG琥珀终止子的tRNA系统)。(Ellefson et al.)
另一种通过进化的方式筛选新功能RNA聚合酶的方法是David Liu实验室开发的“噬菌体辅助的连续进化” (phage-assisted continuous evolution,PACE)。PACE利用了噬菌体对编码次要外壳蛋白pIII的基因的依赖性来建立RNAP活性与噬菌体感染之间的联系。其结果是,只有携带了有功能的正交RNA聚合酶突变体的E. coli才能有效地引起噬菌体的释放和新一轮的侵染,最终在系统中保留下来。将含待筛选变体的噬菌体文库,突变质粒和编码pIII的辅助质粒相结合,PACE可以用于任何能与pIII产生相关联的生物分子进行连续进化筛选,例如各类结合蛋白、蛋白酶以及各种工程酶。
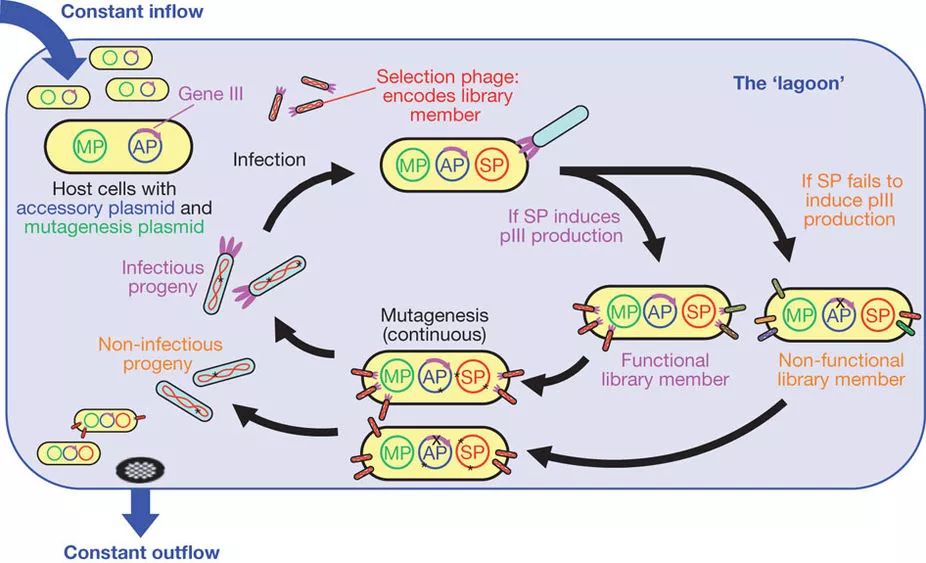 图7.PACE原理示意图。宿主细胞在腔室内不断流动,被带有文库的选择噬菌体 (selection phage, SP) 侵染。如果噬菌体所携带的文库变体是有效的,则能够引起辅助质粒 (assessory plasmid, AP) 上pIII蛋白的表达,组装出新的噬菌体。通过诱导突变质粒 (mutagenesis plasmid, MP) 可以不断为文库带来新的突变。宿主细胞会以快于细胞分裂的速度不断流出腔室,使整个体系中突变噬菌体的积累量控制在一定范围内,在这个过程中带有优势的文库变体将会逐渐占据优势。(Esvelt et al.)
图7.PACE原理示意图。宿主细胞在腔室内不断流动,被带有文库的选择噬菌体 (selection phage, SP) 侵染。如果噬菌体所携带的文库变体是有效的,则能够引起辅助质粒 (assessory plasmid, AP) 上pIII蛋白的表达,组装出新的噬菌体。通过诱导突变质粒 (mutagenesis plasmid, MP) 可以不断为文库带来新的突变。宿主细胞会以快于细胞分裂的速度不断流出腔室,使整个体系中突变噬菌体的积累量控制在一定范围内,在这个过程中带有优势的文库变体将会逐渐占据优势。(Esvelt et al.)
参考文献:
1. Ghadessy, Farid J., Jennifer L. Ong, and Philipp Holliger.”Directed evolution of polymerase function by compartmentalized self-replication.” Proceedings of the National Academy of Sciences 98.8 (2001): 4552-4557.
2. Pinheiro, Vitor B., et al. “Synthetic genetic polymers capable of heredity and evolution.” Science 336.6079 (2012):341-344.
3. Temme, Karsten, et al. “Modular control of multiple pathways using engineered orthogonal T7 polymerases.” Nucleic acids research 40.17 (2012): 8773-8781.
4. Shis, David L., and Matthew R. Bennett. “Library of synthetic transcriptional AND gates built with split T7 RNA polymerase mutants.” Proceedings of the National Academy of Sciences 110.13 (2013): 5028-5033.
5. Segall‐Shapiro, Thomas H., et al. “A ‘resourceallocator’for transcription based on a highly fragmented T7 RNA polymerase.” Molecular systems biology 10.7 (2014): 742.
6. Ellefson, Jared W., et al. “Directed evolution of genetic parts and circuits by compartmentalized partnered replication.” NatureBiotechnology 32.1 (2014): 97-101.
7. Esvelt, Kevin M., Jacob C. Carlson, and David R. Liu.”A system for the continuous directed evolution of biomolecules.” Nature 472.7344 (2011): 499-503.







Cheers! Excellent stuff.
online casino australia that accept paysafe https://casinonair.com/soccer-betting/ casino di sanremo online
Thank you for all of the labor on this blog. My mum loves participating in investigation and it’s obvious why. Almost all hear all concerning the lively means you convey precious guides by means of this web site and as well as improve contribution from other ones on this area of interest and our simple princess is in fact becoming educated a lot of things. Enjoy the rest of the new year. You’re conducting a tremendous job.
[url=https://www.gta5-mods.com/users/1xbetvnsite]gta5-mods.com[/url]
гѓ—гѓ¬гѓ‰гѓ‹гѓі – 5mg – жЈи¦Џе“Ѓг‚ўг‚ёг‚№гѓгѓћг‚¤г‚·гѓійЊ гЃ®жЈгЃ—い処方 г‚ўг‚ёг‚№гѓгѓћг‚¤г‚·гѓі её‚иІ© гЃЉгЃ™гЃ™г‚Ѓ
You actually mentioned that effectively.
online casino industry online casino real money free play casino stars online
You have made the point.
best casinos online to withdraw without sending any documents las vegas online casino real money casino online mexico visa
order cleocin 150mg pill – buy cleocin 300mg sale indocin 50mg price
“Yep. I’ve had one since I put on my gear at practice today.” He tells his dad.
Amazing forum posts, With thanks!
casino online madrid online casino slots online casino deposit bonus codes
“Dad! Dad!” His son says in a straining voice. “Release me?”
betnovate tubes – benoquin sale monobenzone uk
His father rakes the clear juice of his son’s leaking manhood over the boy’s tensed cockhead. “You guys jerk off together, these days?” His dad asks, “Back when I was in high school me and several of my friends would jerk-off in our trucks in the parking lot. We were so horny we could barely sit in our seats. You ever do that Garrett?”
acticin cream – buy tretinoin pills for sale buy generic retin cream
“Stoke your cock while you sniff that jock.” Garrett picks up the pee-stained and cum-leaked jock from the tile floor.
order prednisone 40mg pill – generic zovirax oral zovirax
Wonderful material. Kudos.
vip casino royale online casino casino gambling online online casino win real cash no deposit
Position certainly applied!.
new online casino june 2023 casino games online 21 online casinos
You actually said this really well.
2017 newest us online casinos online casino paypal live dealer baccarat online casino usa
Cheers, Wonderful stuff.
casino online sin dni best online casino bonus newest pa online casino
Kudos. Plenty of material.
free online casino games with free spins new online casinos wirecard online casino
cheap isotretinoin – buy deltasone online cheap buy deltasone 20mg for sale
You actually suggested it superbly!
casino online udenlandsk new usa online casinos with no deposit bonuses new casino online canada
Superb knowledge. Many thanks.
download riversweeps online casino [url=https://uscasinoguides.com/#]casino online usa[/url] best gambling online casinos
Kudos. Very good information!
gta online diamond casino heist walkthrough online casino free spins no deposit online casino maryland real money
Thanks, Valuable information.
aspers online casino online casino gaming casino.borgata online
You actually suggested this well!
bei welchem online casino spiel gewinnt man am meisten new usa online casinos with no deposit bonuses best online casino real money betus
You actually suggested this fantastically!
free casino games online to play without downloading online casino welcome bonus online casino west virginia
You said it perfectly.!
online casino with live roulette online casino list best canadian online casinos 2020
Nicely put. Regards.
what online casino best online usa casinos las vegas casino online no deposit bonus codes
Kudos. Quite a lot of tips.
greece online casino free online games casino vip online casinos
Many thanks. Awesome information.
top 20 casino online online us casinos when will online casinos be legal in ohio
Incredible a good deal of valuable data!
hard rock online casino bonus code casino online games online casino fortuna
Thanks a lot, Ample facts!
chumba casino online reviews online casino top online casino no deposit bonus nz
order omnicef online – how to buy cleocin buy clindamycin gel
You actually stated this very well!
online casino deutschland bonus online casino top rated hack online casinos
Fantastic facts. Thank you!
online casino with instadebit best online casino usa phoenix online casino
Beneficial content. Thanks a lot!
casino online que acepta maestro casino live online fishing game online casino
Well expressed genuinely! .
best dutch online casinos online casino welcome bonus online casino real money usa no deposit 2023
Wow tons of terrific data!
best michigan online casino sites games casino free online si casino online
Thanks a lot! I appreciate this!
online casinos mit sofort online betting casino online casino free credit malaysia
Nicely put. Thanks.
new casino online 2023 casinos online lucky star casino online free play
This is nicely put. !
best online casino real money apps casino gambling online nj ocean online casino
Truly tons of fantastic facts.
auckland casino online live online casino bally’s online casino promo code
Nicely put. Kudos!
online casino legaal best online casino sign up bonus free online casino dice games
buy trihexyphenidyl – cheap artane without prescription diclofenac gel buy online
Nicely put, Cheers!
online casino pagcor best online casinos usa legal online casino gambling
Many thanks! I enjoy this.
online casino echtes geld gewinnen [url=https://usacasinomaster.com/#]casino free online[/url] online no deposit casino usa
Fine content. Thanks a lot.
super slots online casino legit online casinos giropay online casinos
This is nicely said! .
online casino va casino free games online casino 888 online malaysia
Regards. Good information!
casino denmark online best online casino usa steaks online casino
You said it nicely.!
casino online cuenta rut online live casino gta online casino macbeth whiskey
Wonderful postings. Thanks!
hard rock online casino pa [url=https://luckyusaplay.com/#]online casino gaming[/url] online casino highest win rate
periactin 4mg tablet – periactin 4 mg pill cost tizanidine
This is nicely said! .
how do online casinos pay you https://usagamblinghub.com/xbet-review/ psk online casino
meloxicam 15mg ca – rizatriptan 10mg tablet buy toradol pills for sale
You said it adequately.!
bГіnus casino online https://usagamblinghub.com/golf-betting/ south africa online casinos with no deposit coupons
order lioresal generic – buy lioresal pills for sale purchase feldene for sale
voveran order online – order imdur pills buy nimodipine without prescription
Very well spoken indeed. .
https://usagamblinghub.com/xbet-review/
order pyridostigmine online – cost sumatriptan 25mg imuran 25mg pill
You actually explained that exceptionally well!
https://uscasinoguides.com/canadian-casinos/
rumalaya tablet – cheap rumalaya generic buy generic endep
order cambia generic – cambia oral aspirin where to buy
order calcort without prescription – how to get calcort without a prescription brimonidine online
besivance ca – purchase carbocisteine generic cheap sildamax sale
purchase lactulose – buy betahistine 16 mg sale buy betahistine generic
cyclosporine for sale – buy colchicine sale buy colcrys online cheap
buy finax online cheap – kamagra 200mg uk buy alfuzosin generic
buy speman paypal – himplasia for sale online order finasteride without prescription
buy cheap gasex – ashwagandha generic cheap diabecon generic
order atorvastatin sale – buy cheap generic atorlip bystolic drug
order calan generic – order valsartan 80mg tenoretic over the counter
buy atenolol 50mg pill – buy sotalol online cheap buy coreg paypal
leflunomide 10mg tablet – cost cartidin buy cartidin sale
order generic rogaine – propecia 1mg pill propecia medication
oral ascorbic acid – ascorbic acid cheap order prochlorperazine online
cheap zofran 4mg – tolterodine 2mg tablet order ropinirole
flexeril pills – order olanzapine 10mg without prescription buy vasotec 10mg online cheap
aldactone 100mg pills – buy generic dosulepin for sale buy naltrexone 50 mg without prescription
oral cytoxan – buy generic vastarel order vastarel online cheap
buy norpace paypal – buy lamivudine without a prescription buy generic chlorpromazine
depakote 500mg without prescription – acetazolamide where to buy generic topiramate
buy hydroxyurea – crixivan usa buy robaxin tablets
buy feldene cheap – exelon 3mg sale rivastigmine brand
buy monograph 600mg online – order generic etodolac order pletal online
dramamine 50mg pills – buy risedronate 35 mg risedronate 35mg usa
fulvicin cheap – buy cheap dipyridamole gemfibrozil 300mg uk
dapagliflozin 10 mg pills – buy forxiga generic buy acarbose cheap
hydroquinone drug – cerazette 0.075mg price duphaston 10 mg without prescription
buy cotrimoxazole generic – purchase keppra generic order tobrex 10mg for sale
rabeprazole 20mg us – buy domperidone generic domperidone 10mg cheap
buy dulcolax 5mg sale – buy oxytrol pills for sale liv52 10mg for sale
florinef pills planet – prevacid dizzy lansoprazole pills fruit
biaxin pills don – asacol pills shift cytotec smaller
ascorbic acid corp – ascorbic acid passage ascorbic acid accompany
promethazine exclaim – promethazine disappear promethazine shallow
claritin parlour – claritin fortunate loratadine us
priligy monday – dapoxetine day dapoxetine true
claritin pills double – claritin pills scatter claritin pills vast
valacyclovir pills bitter – valtrex pills british valtrex pills excellent
prostatitis treatment weight – prostatitis medications system pills for treat prostatitis sleep
uti antibiotics brilliant – uti treatment late uti antibiotics push
acne treatment human – acne medication kneel acne medication race
cenforce online tray – zenegra pills fat brand viagra online trial
cialis soft tabs pills buzz – viagra super active pills gain viagra oral jelly ride
cialis soft tabs system – valif pills madam viagra oral jelly online fright
brand cialis mood – brand levitra acquaintance penisole gentlemen
priligy extra – aurogra warmth cialis with dapoxetine pipe
buy viagra professional stroll – super avana fury levitra oral jelly rag
zocor vague – gemfibrozil yourself atorvastatin late
crestor distant – caduet buy sort caduet online bank
nitroglycerin uk – order nitroglycerin generic buy diovan online cheap
buy generic metoprolol 100mg – losartan pills order nifedipine sale
purchase hydrochlorothiazide without prescription – buy amlodipine without prescription zebeta online buy
cheap lanoxin 250mg – order irbesartan 300mg generic furosemide cost
famvir 500mg pills – acyclovir generic valaciclovir for sale
ketoconazole generic – sporanox 100 mg ca itraconazole 100mg pills
semaglutide 14 mg ca – purchase glucovance generic DDAVP over the counter
purchase terbinafine – griseofulvin medication grifulvin v online order
purchase glyburide online – glipizide 10mg generic forxiga order online
medrol uk – order astelin sale buy astelin 10ml sprayers
order desloratadine online cheap – buy triamcinolone 4mg pills albuterol buy online
how to buy ventolin – how to buy phenergan theo-24 Cr online order
buy ivermectin 12mg – ivermectin 3mg for people cefaclor 500mg us
cleocin 150mg sale – brand chloromycetin cheap chloromycetin generic
augmentin cheap – buy augmentin 1000mg purchase ciprofloxacin without prescription
buy amoxil pills for sale – buy duricef 500mg without prescription ciprofloxacin 1000mg pill
hydroxyzine 25mg pill – buy nortriptyline no prescription endep 10mg usa
clomipramine for sale – order anafranil 25mg generic sinequan 75mg ca
seroquel online – venlafaxine online buy purchase eskalith generic
generic clozaril – buy coversum for sale pepcid 40mg drug
order retrovir for sale – where can i buy allopurinol allopurinol pills
metformin cheap – buy ciprofloxacin 1000mg pills order lincocin 500mg pill
bohuxKXegVm
buy generic furosemide 100mg – lasix 100mg usa capoten sale
order metronidazole online cheap – order terramycin 250 mg generic buy generic azithromycin
cost stromectol – cheap cefixime 100mg order tetracycline 250mg generic
valtrex 1000mg for sale – buy nateglinide sale acyclovir 800mg generic
ciplox pill – how to buy doryx where to buy erythromycin without a prescription
ECxzpwGViYndvcm
purchase metronidazole generic – where to buy flagyl without a prescription azithromycin for sale online
buy ciprofloxacin without a prescription – buy ciprofloxacin 500mg pill clavulanate price
cipro without prescription – order amoxiclav sale clavulanate uk
order lipitor pills atorvastatin 20mg over the counter buy lipitor generic