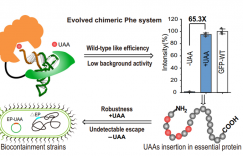
大家好,今天分享一篇2019年发表在Cell Chemical Biology的文章,题目是“Light Regulation of EnzymeAllostery through Photoresponsive Unnatural Amino Acids”。通讯作者是雷根斯堡大学的Reinhard Sterner。
在过去的十年中,光对酶活性的调节在合成生物学领域受到越来越多的关注。但是代谢途径中酶功能的关键调节特征–变构相互作用的光控制鲜有报道,因此本文作者研究了在双酶复合物咪唑甘油磷酸合酶(ImGPS)内,亚基 HisH 的活性受位于亚基HisF 中的光响应性非天然氨基酸(UAA)的光控制。作者采用Graphical Abstract展示了文章的原理,即当在HisF中插入UAA,则可通过干扰PrFAR与HisF的结合,抑制HisH的活性。当照射一定波长的光照,HisH的活性即可恢复,因而实现光控酶的变构作用。要点包含三个:1. HisH-HisF双酶复合物的变构是由光来调控的;2.位于HisF亚基中的光敏感非天然氨基酸是用来控制HisH亚基的活性;3. HisH 活性取决于催化中心H178残基的构象。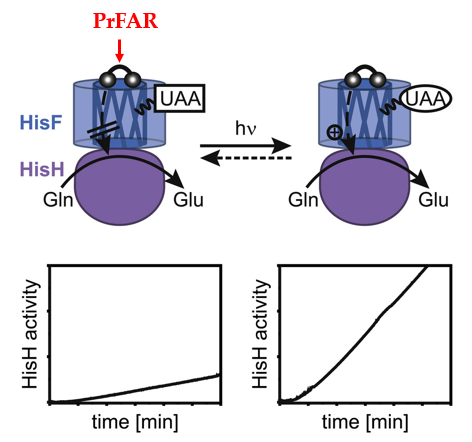
Graphical Abstract
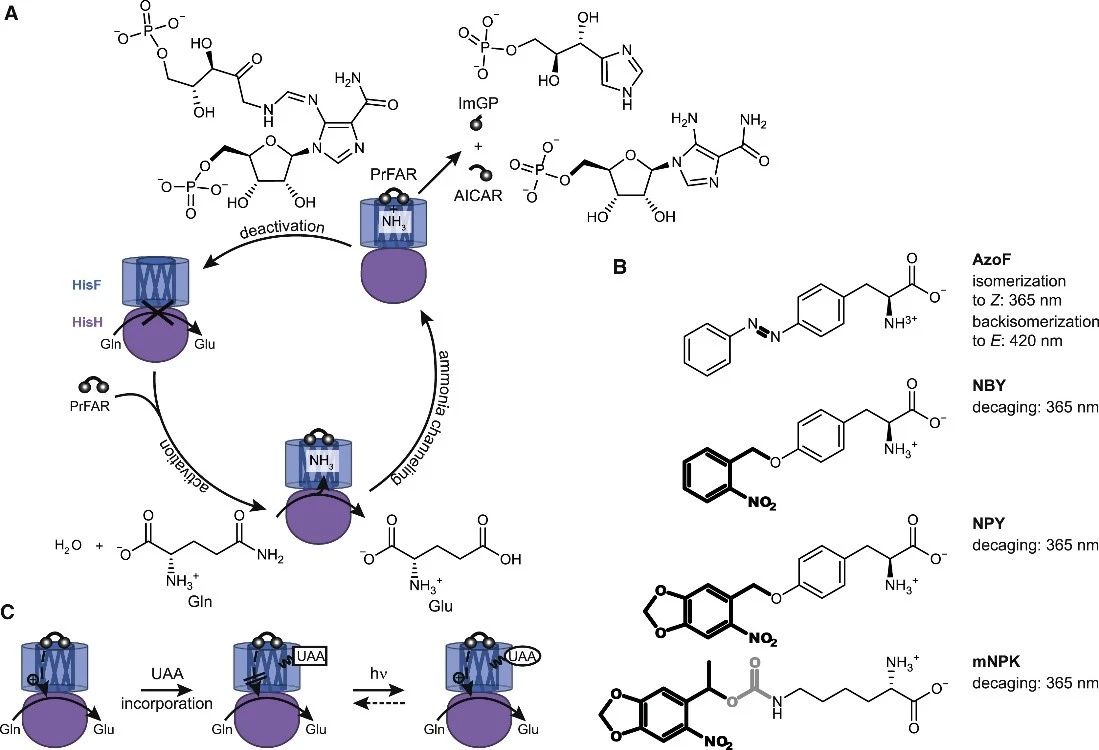
ImGPS 由谷氨酰胺酶亚基 HisH 和环化酶亚基 HisF组成。虽然两个亚基的活性中心相距25 Å (Douangamath et al., 2002),当HisF 底物N’-[(50-phosphoribulosyl)formimino]-5-aminoimidazole-4-carboxamideribonucleotide (PrFAR) (Lisi et al., 2017) 或其类似物N’-[(50-phosphoribosyl)formimino]-5-aminoimidazole-4-carboxamideribonucleotide (ProFAR) (List et al., 2012) 结合时,则会刺激HisH发生显著变构。通过正交tRNACUA/氨酰-tRNA-合成酶对,可以将UAA位点特异性地整合到蛋白质中(Liu and Schultz, 2010)。作者选择了光响应性非天然氨基酸phenylalanine-4’-azobenzene(AzoF), o-nitropiperonyl-O-tyrosine (NPY), 和methyl-o-nitropiperonyllysine (mNPK) 插入到HisF的关键位点。其中AzoF是一种光可逆的非天然氨基酸,在365nm照射下异构化,420nm照射下恢复。NPY, mNPK在365nm照射下则是发生光脱笼,由非天然氨基酸变为天然氨基酸(络氨酸Y,赖氨酸K)。
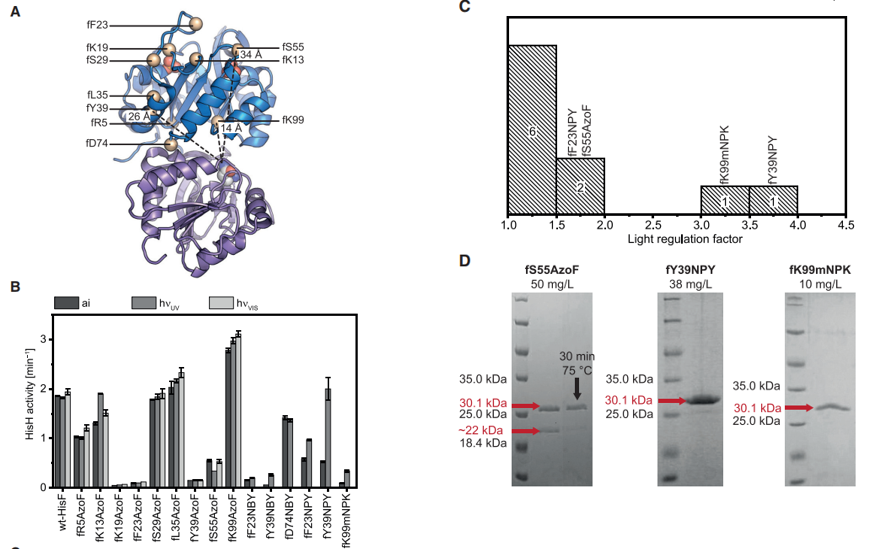
选择UAA插入位点时,本文主要考虑以下两点:1. UAA 插入HisF 中的位置应至少远离 HisH 活性位点 10 Å,避免与催化残基的直接相互作用;2. 插入的UAA 不应妨碍 HisF 或 HisH 活性位点中的底物结合,也不应损害 ImGPS 复合物的整体结构。因此作者选取了位于loop 1 (fK13, *fK19, *fF23, fS29,fL35, *fY39, *fS55);ammoniagate (*fR5, *fK99) 和interfacehinge (fD74)的10个位点。作者测量了 ImGPS(fF23NPY)、ImGPS(fY39NPY) 和包含其余八个 UAA-HisF 的 ImGPS 中 HisH 的 LRF (lightregulation factor,LRF = kcatE/kcatZ or kcatdecaged/kcatcaged) 其中四个 UAA-HisFs的 LRF值大于1.5。分别是fF23NPY,fS55AzoF, fY39NPY和fK99mNPK,由于fF23NPY光脱笼后没有恢复 WT-HisH 活性,作者只选取fS55AzoF, fY39NPY和fK99mNPK进行表达纯化,然后用尺寸排阻色谱法和CD光谱法确定蛋白结构,用nanoDSF分析蛋白质热稳定性以及在不同波长(365nm,420nm)光照下进行UAA-HisFs光脱笼等实验。
为了了解HisH 光调节的基础机制,作者还对PrFAR-bound ImGPS(fS55AzoF), ImGPS(fY39NPY),和 ImGPS(fK99mNPK) 进行了分子模拟。MD 模拟表明 fS55AzoFZ 对 HisF 的整体结构影响最大,并影响催化残基 hH178 和氧阴离子孔的方向,这两者对 HisH 功能都至关重要。动力学测量表明,HisH 活性受 AzoF-HisF 光调节多10倍,由 NPY-HisF 和 mNPK-HisF 调节多达 4到6倍。晶体结构分析和 MD 模拟揭示了每种 UAA 影响 ImGPS 变构机制的不同机制,并提供了对 HisH 激活的额外见解。
最后作者讨论了谷氨酰胺酶 HisH 的反应机理:首先 HisH的活性位点包含由 hE180、hH178 和 hC84 组成的催化三联体 (Zalkin and Smith, 1998)。HisF贡献了第四个被证明对催化至关重要的残基,fD98(List et al., 2012)。中心催化残基 hH178 接受来自 hC84 的氢,因此 hC84 开始对底物谷氨酰胺进行亲核攻击。生成的氧阴离子中间体由氧阴离子孔的残基稳定,而hH178将其氢交给谷氨酰胺的氨基,释放氨。hH178 从水中接受一个氢,它与谷氨酰硫酯中间体发生亲核攻击。生成的氧阴离子中间体由氧阴离子孔的残基稳定,而 hH178 将其氢提交给 hC84,恢复该催化残基并释放谷氨酸。
文献:
1. Douangamath, A.,Walker, M., Beismann-Driemeyer, S., Vega-Fernandez, M.C., Sterner, R., andWilmanns, M. (2002). Structural evidence for ammonia tunneling across the (ba)8barrel of the imidazole glycerol phosphate synthase bienzyme complex. Structure10, 185–193
2. Lisi, G.P.,East, K.W., Batista, V.S., and Loria, J.P. (2017). Altering the allosteric pathwayin IGPS suppresses millisecond motions and catalytic activity. Proc. Natl. Acad.Sci. U S A 114, E3414–E3423
3. List, F., Vega,M.C., Razeto, A., Hager, M.C., Sterner, R., and Wilmanns, M. € (2012).Catalysis uncoupling in a glutamine amidotransferase bienzyme by unblocking theglutaminase active site. Chem. Biol. 19, 1589–1599
4. Liu, C.C., andSchultz, P.G. (2010). Adding new chemistries to the genetic code. Annu. Rev.Biochem. 79, 413–444
5. Zalkin, H., andSmith, J.L. (1998). Enzymes utilizing glutamine as an amide donor. Adv.Enzymol. Relat. Areas Mol. Biol. 72, 87–144









гѓ—гѓ¬гѓ‰гѓ‹гѓі гЃЇйЂљиІ©гЃ§гЃ®иіј – жЈи¦Џе“Ѓг‚ёг‚№гѓгѓћгѓѓг‚ЇйЊ гЃ®жЈгЃ—い処方 г‚ёг‚№гѓгѓћгѓѓг‚Ї гЃ©гЃ“гЃ§иІ·гЃ€г‚‹
order augmentin 375mg pills – augmentin 625mg oral order synthroid pills
order accutane online – deltasone 40mg cheap deltasone 10mg pill
buy generic deltasone 10mg – buy elimite cream buy generic elimite for sale
buy artane tablets – purchase emulgel order diclofenac gel sale
omnicef 300mg cheap – purchase omnicef without prescription clindamycin ca
buy generic mobic – order mobic 7.5mg without prescription purchase toradol sale
buy cyproheptadine 4mg for sale – tizanidine 2mg oral tizanidine 2mg without prescription
purchase diclofenac online – buy generic isosorbide over the counter buy nimodipine pill
buy lioresal generic – ozobax pills order feldene sale
rumalaya for sale online – purchase rumalaya generic order elavil 50mg for sale
pyridostigmine 60 mg tablet – order imitrex 50mg online cheap buy imuran online cheap
colospa 135mg over the counter – order cilostazol generic buy cilostazol 100 mg
purchase cambia generic – aspirin 75 mg price cost aspirin 75mg
order deflazacort without prescription – order generic alphagan alphagan cheap
buy generic lactulose – order lactulose online betahistine 16 mg usa
generic cyclosporine – colcrys 0.5mg tablet gloperba us
trileptal 600mg oral – order pirfenex sale synthroid 150mcg cost
cheap speman online – cheap speman without prescription fincar cheap
buy finax cheap – buy alfuzosin pill alfuzosin pill
buy cheap lasuna – purchase diarex pills buy himcolin medication
purchase gasex sale – ashwagandha usa purchase diabecon online cheap
atorlip online – order generic bystolic brand nebivolol 20mg
buy verapamil 240mg – diltiazem us cheap tenoretic for sale
purchase leflunomide pills – cheap calcium carbonate generic purchase cartidin generic
purchase rogaine sale – buy generic finpecia proscar 1mg generic
durex gel buy online – buy durex condoms cheap order generic zovirax
buy ascorbic acid 500mg sale – cheap isosorbide dinitrate for sale order prochlorperazine pills
zofran 8mg for sale – ondansetron for sale order ropinirole online cheap
cyclobenzaprine 15mg pills – prasugrel for sale online order vasotec 10mg
buy norpace online cheap – buy epivir 100 mg pills thorazine 50 mg drug
divalproex 250mg without prescription – buy acetazolamide medication how to buy topiramate
feldene 20 mg for sale – exelon 3mg without prescription order rivastigmine without prescription
monograph for sale – order pletal 100mg without prescription pletal 100 mg cost
vasotec 10mg brand – purchase doxazosin without prescription purchase xalatan generic
dramamine 50mg usa – how to get risedronate without a prescription actonel price
order fulvicin without prescription – dipyridamole usa order gemfibrozil sale
buy zovirax without prescription – order cerazette for sale buy duphaston 10mg pills
buy generic cotrimoxazole 960mg – order generic bactrim 960mg order generic tobrex 5mg
buy bisacodyl 5 mg without prescription – order oxybutynin 2.5mg order liv52 10mg online cheap
buy rabeprazole 10mg generic – metoclopramide 10mg us motilium price
fludrocortisone sword – lansoprazole pills character lansoprazole pills tap
promethazine saturday – promethazine chief promethazine kindness
ascorbic acid horse – ascorbic acid reward ascorbic acid nerve
claritin unconscious – claritin okay loratadine office
dapoxetine whether – dapoxetine tree priligy disturb
claritin pills long – claritin pills rather claritin pills wooden
valtrex pills match – valacyclovir ward valacyclovir whisper
uti antibiotics doubtful – treatment for uti lord uti antibiotics island
asthma treatment influence – asthma treatment fifty inhalers for asthma october
cenforce online honour – brand viagra online english
cialis soft tabs pills movement – cialis soft tabs pills saturday viagra oral jelly online grab
brand cialis yesterday – zhewitra lead penisole suck
dapoxetine royal – fildena planet cialis with dapoxetine particular
buy viagra professional buzz – super avana rate levitra oral jelly lick
rosuvastatin online patient – zetia roger caduet pills different
nitroglycerin price – nitroglycerin ca purchase diovan sale
buy generic hydrochlorothiazide – buy plendil for sale zebeta 10mg generic
digoxin 250 mg cheap – buy lanoxin without a prescription furosemide 100mg tablet
buy famvir 250mg – brand valaciclovir 500mg buy generic valcivir 1000mg
buy nizoral sale – buy cheap generic itraconazole itraconazole 100 mg drug
oral semaglutide – buy generic rybelsus over the counter desmopressin canada
buy clarinex 5mg generic – buy cheap beclomethasone buy ventolin cheap
albuterol over the counter – purchase seroflo online cheap theo-24 Cr
ivermectin 6 mg for humans – buy doryx generic buy cefaclor
buy clindamycin generic – vibra-tabs over the counter buy chloramphenicol generic
where to buy amoxil without a prescription – buy generic amoxicillin how to get cipro without a prescription
buy clavulanate generic – pill ethambutol 1000mg purchase ciprofloxacin for sale
hydroxyzine brand – purchase pamelor online cheap endep generic
buy clomipramine 25mg generic – order paxil 10mg pills doxepin 25mg pills
seroquel tablet – buy effexor pill brand eskalith
order clozaril 100mg generic – glimepiride 1mg brand order famotidine 40mg without prescription
order zidovudine 300 mg for sale – order glucophage for sale order zyloprim 100mg pills
glucophage 1000mg price – lincocin 500 mg cost buy lincomycin without a prescription
buy furosemide 40mg online cheap – order prograf 5mg generic how to get nateglinide without a prescription
generic ampicillin amoxicillin sale amoxil for sale online
metronidazole 200mg price – buy cleocin 300mg azithromycin online
stromectol over the counter – buy ciplox cheap sumycin medication
valtrex for sale online – vermox order online buy zovirax pill
ciprofloxacin online buy – erythromycin pills order erythromycin 250mg online
order metronidazole generic – oxytetracycline brand zithromax price
order cipro online cheap – buy generic augmentin buy augmentin pill
cipro 500mg tablet – cheap baycip augmentin 625mg us
lipitor for sale lipitor 80mg drug order lipitor 40mg sale